はじめに
この記事では、SciPyライブラリのndimage.percentile_filterを使用して画像の平滑化を行う方法について解説します。パーセンタイルフィルタの基本概念から実装方法、さまざまなパラメータの効果まで詳細に説明しています。
コード&解説
モジュールのインポート
バージョン
画像の読み込み
サボテンのすみれ丸の画像をplt.imread()関数で読み込みます。
グレースケール変換
skimage.color モジュールの rgb2gray 関数を使用して RGB 画像をグレースケール画像に変換します。変換した画像を cmap=”bone” パラメータで表示すると、以下のようになります。

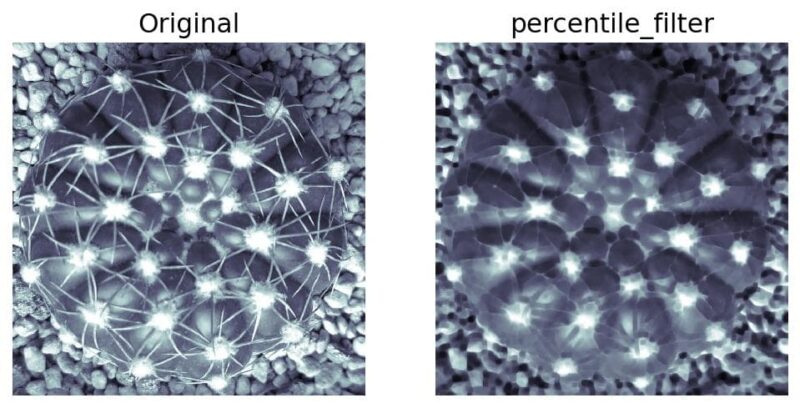
パーセンタイルフィルタ
パーセンタイルフィルタのパーセンタイルを10、サイズを10として処理すると、以下のような画像が得られます。処理後の画像では高輝度部分である棘が目立たなくなり、まるで棘のないサボテンのように見えることがわかります。
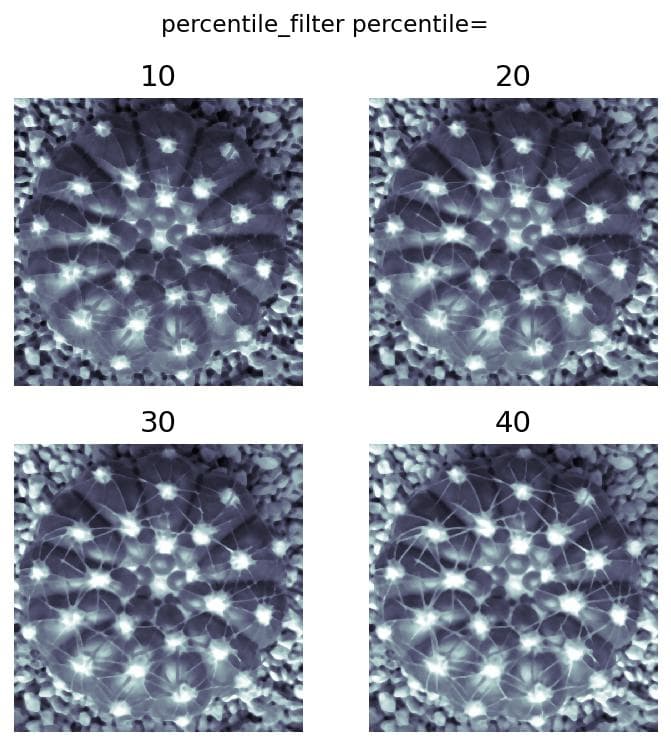
percentileを変化させた場合
パーセンタイル値を大きくするにつれて、画像の鮮明さが増していく様子が観察できます。
sizeを変化させた場合
フィルタのサイズを大きくするにつれて、画像はより一層ぼやけていきます。

footprintを変化させた場合
footprintパラメータを使用してフィルタをかける範囲を変更できます。この例では、scikit-imageの
morphologyモジュールを活用して、フィルタの適用範囲を設定しました。結果は以下の通りです。
Generate footprints (structuring elements) — skimage 0.26.0 documentation

まとめ
SciPyのndimage.percentile_filterを使った画像平滑化は、ノイズ除去や画像の特徴強調に効果的です。パーセンタイル値を調整することで、メディアンフィルタよりも柔軟な画像処理が可能になります。この方法はエッジ保存や特定のノイズ特性に対応する画像処理タスクに特に有用です。
参考
percentile_filter — SciPy v1.17.0 Manual


コメント