はじめに
scikit-image(skimage)のexposure.histogram関数を使用して画像のヒストグラムを作成する方法について解説します。画像のヒストグラムとは、画像内の各ピクセル値の分布を可視化したもので、画像処理や分析において重要なツールです。
解説
モジュールのインポートなど
バージョン
画像の読み込み
パロディア属スミレ丸の画像を読み込みました。img_as_float関数でデータ範囲を0-1に正規化し、rgb2gray関数によりグレースケール画像に変換しました。読み込んだ画像は以下のように表示されます。

ヒストグラムデータの作成
exposure.histogram
ヒストグラム作成には、処理したい画像とヒストグラム計算に使用するビンの数を指定します。
np.histogram
ヒストグラム作成には、対象とする画像とビンの数を指定します。ビンの範囲は、画像の最小値から最大値の間に自動的に設定されます。
plt.hist (ax.hist)
exposure.histogram関数を使用する際は、ヒストグラム化したい画像と計算に使用するビンの数を指定します。なお、画像データは1次元配列に変換する必要があります。その他のパラメータや動作はnp.histogram関数と同様です。
binの比較
skimage.exposure.histogramのビンの形状は10であるのに対し、numpyとmatplotlibのヒストグラムのビンは11となります。この違いは、skimageのヒストグラムのビンの値がビンの中心を表しているためです。一方、numpyとmatplotlibはビンのエッジを返すため、形状が11になります。
頻度の比較
頻度の値については、skimage, numpy, matplotlibで同じとなります。
ヒストグラムの表示
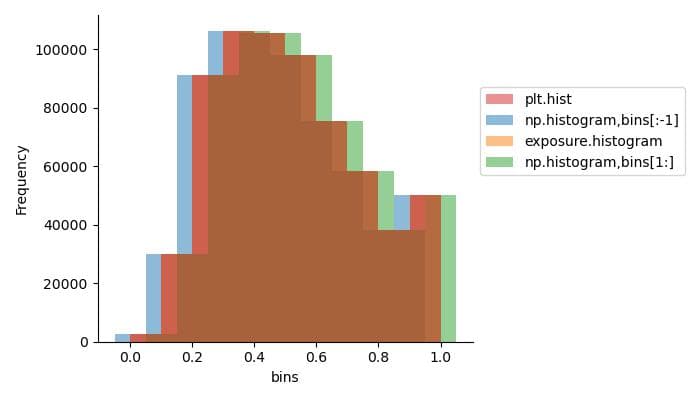
skimage、numpy、matplotlibで作成したヒストグラムデータをmatplotlibのbar関数で表示すると以下のようになります。なお、numpyのヒストグラムを表示する際は、np_bin[:-1]またはnp_bin[1:]を使用して配列の形状を調整する必要があります。
matplotlibのax.hist関数の場合、skimageのヒストグラムと同様に、ビンの中央値でバーを表示するため、matplotlibとskimageのヒストグラム表示は一致します。
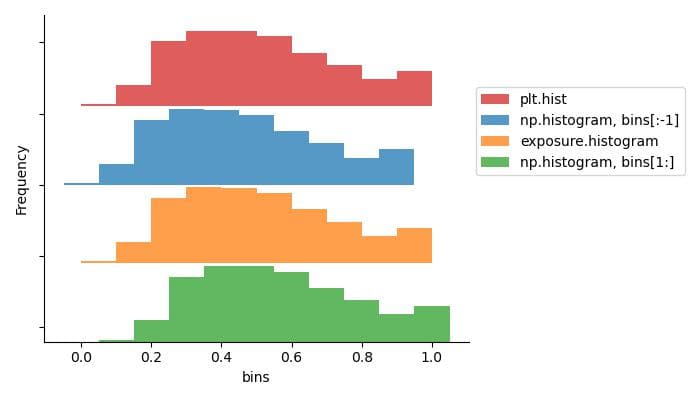
オフセットをかけて表示すると以下のようになります。
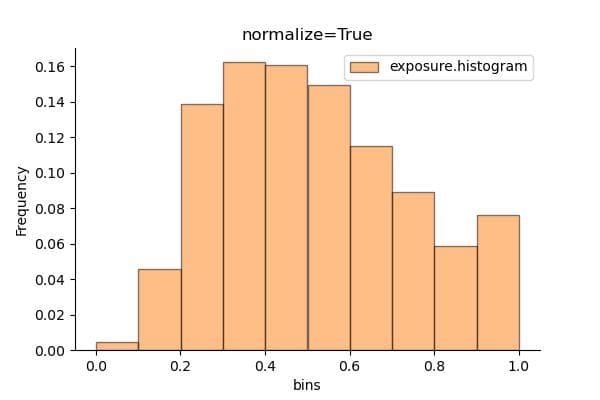
規格化(normalize)
skimageでは、normalize=Trueを設定することでヒストグラムを規格化(正規化)でき、頻度の合計値が1になります。規格化したヒストグラムは以下のようになります。
一方、matplotlibで正規化するには、density=Trueパラメータを設定します。その結果は以下のとおりです。
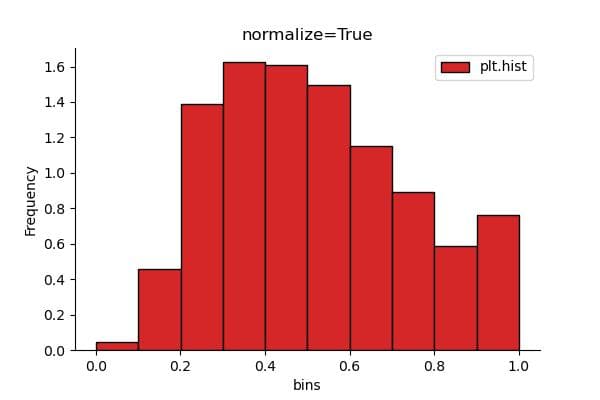
この頻度の合計値はそのままでは1になりません。合計を1にするためには、ビンの幅(np.diff(plt_bins)[0])を掛ける必要があります。
np.histogramの場合も同様となります。
まとめ
scikit-imageのexposure.histogram関数を使うことで、画像のピクセル値分布を簡単に取得・可視化できます。他のライブラリと比較しても使いやすく、画像処理ワークフローに組み込みやすい特徴があります。ヒストグラム分析は画像の特性把握や処理の前処理として非常に有用なテクニックです。

コメント