はじめに
この記事では、SciPyのndimageモジュールに含まれるmeasurements関数とmorphology関数を用いて、実生サボテンの画像処理を行う方法について解説します。具体的には、画像からサボテンを検出し、ラベリングを行った後、それぞれのサボテンのサイズや特徴を計測する手順を示します。これらの技術は、植物の成長モニタリングや自動分類などの応用に役立ちます。
コード & 解説
モジュールのインポート
画像の読み込み
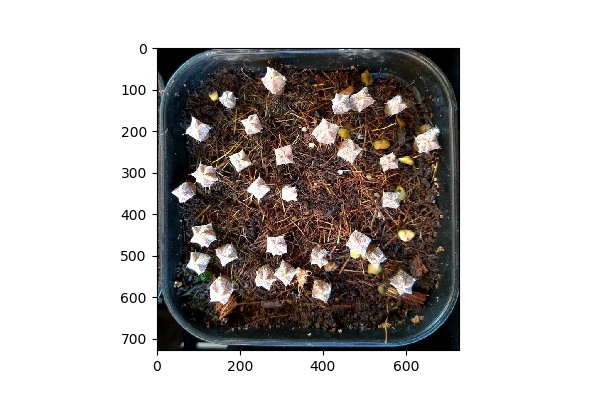
今回使用する画像は、アストロフィツム属のストロンギロゴナムの実生サボテンで、種まきから約8ヶ月経過したものです。
成長すると、下記サイトのような姿になります。

白黒画像で読み込み、その形状とサイズを調べたところ、shape=(727, 727)、size=528529であることがわかった。
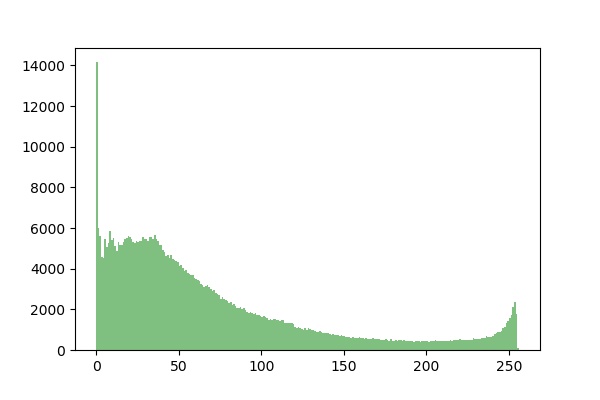
画像のヒストグラムの表示
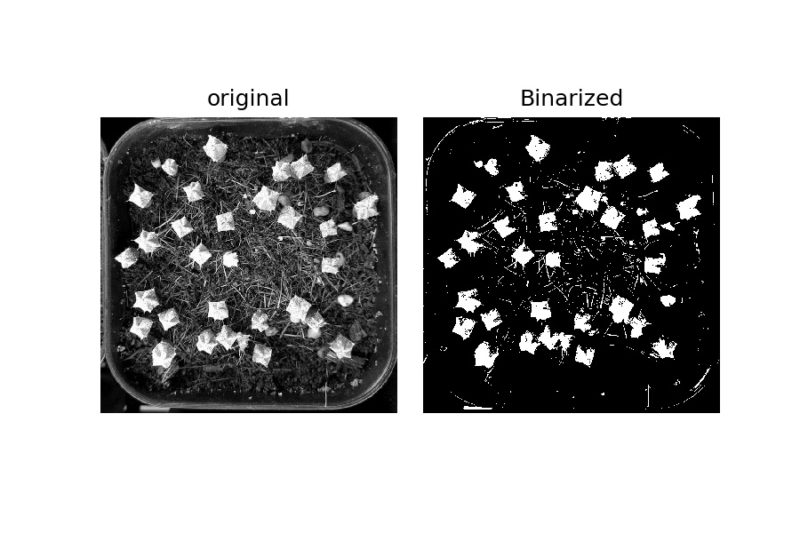
2値化
二値化は、im2 = (im>150)のように処理することで、値が150以上の部分のみTrueとなり、それ以外はFalseとなるデータが得られます。このデータ内のTrue値の数は、im2.sum()で確認でき、65179個あることがわかりました。
二値化した画像は以下のようになります。
画像のラベリング
scipy.ndimage.measurementsモジュールのlabel()関数を使用すると、画像内の白い領域を数えることができます。この関数は、ラベル付けされた領域の画像(labels)とラベル付けされた領域の数(nbr_objects)の2つの値を返します。
二値化画像の白い領域を計測したところ、2195個という結果が得られました。この数値が多いのは、画像中のサボテン以外のノイズも計測しているためであり、正確な分析のためにはノイズ除去が必要です。
binary_openingによる小さい領域の除去
morphology.binary_opening()関数を使用することで、画像中の小さい領域(ノイズ)を除去することができます。iterations引数の値を大きくするほど、より大きな領域が除去されるようになります。
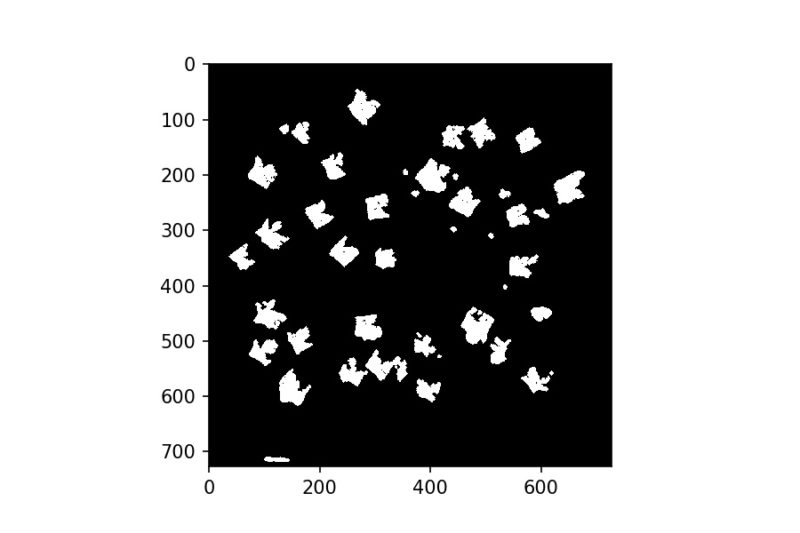
白い領域の数は45となりました。
マスクによる小さい領域の除去
morphology.binary_opening()以外にもノイズを除去する方法として、マスクを用いる手法があります。
measurements.sum()関数を使用すると、ラベル付けされた各白領域のサイズを取得できます。mask = areas > 755とすることで、指定したサイズ以下の領域をFalseとし、mask[labels_op]を適用すれば、755ピクセル未満の小さな白領域(ノイズ)が除外された二値化画像が得られます。
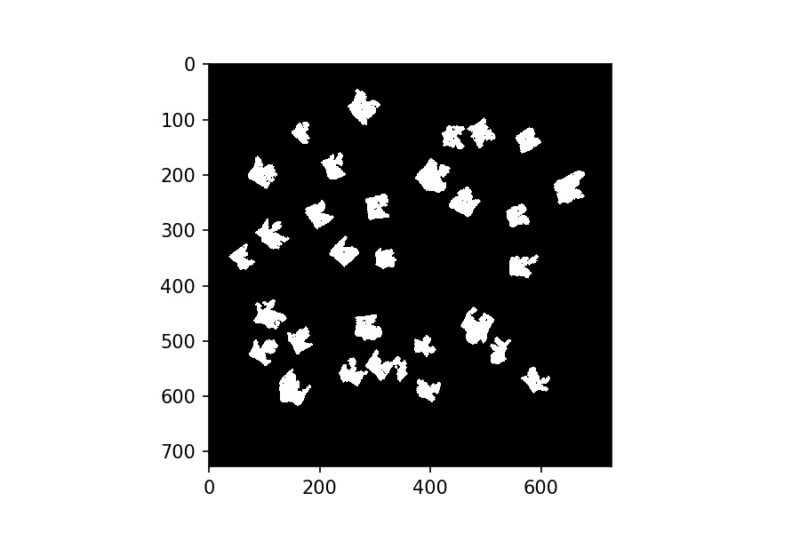
処理の結果、白い領域の個数は30個となりました。これまでの二値化処理の流れをまとめると、以下の図のようになります。

ラベリングされた画像
30個の白い領域がラベリングされた画像を表示します。
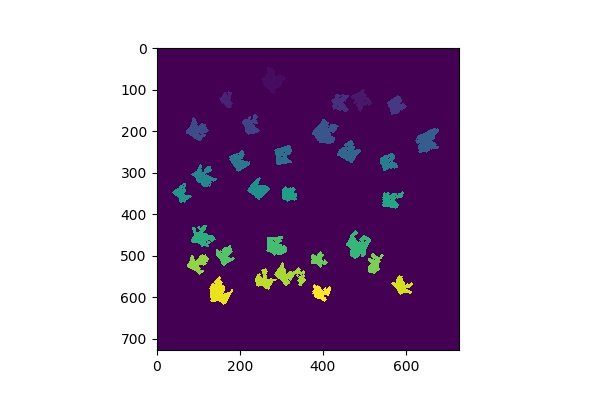
背景が0のため、ラベル値1の位置が分かりにくいです。そこで以下のような処理を行います。
背景値である0の部分をnp.nanに置換すると、その領域は色が表示されなくなるため、以下のようなより明瞭な画像が得られます。
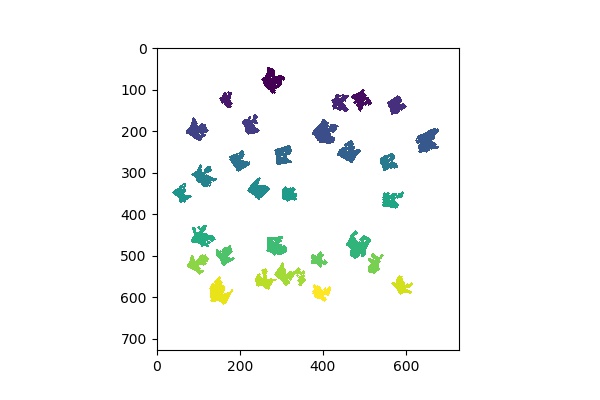
ラベリングされた領域の重心
ラベリングした領域の重心は、measurements.center_of_mass()関数で求めることができます。この重心を画像中に赤点で表示し、さらに各重心の右上にそのインデックスを追記しました。
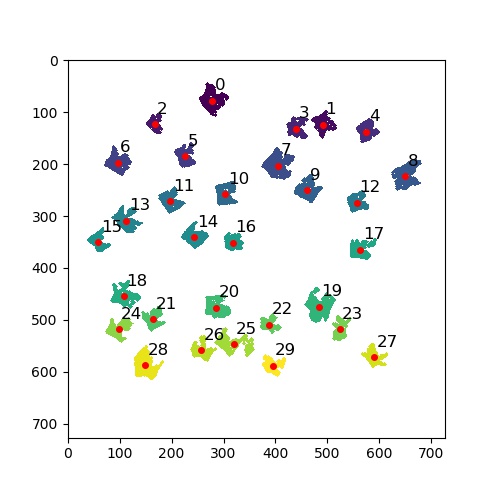
ラベリングした領域の大きさ
ラベリングした各領域のサイズは、measurements.sum()関数を使って個別に計測できます。先ほど求めた重心の位置情報を活用し、各重心の右上に対応する領域のサイズを表示しました。
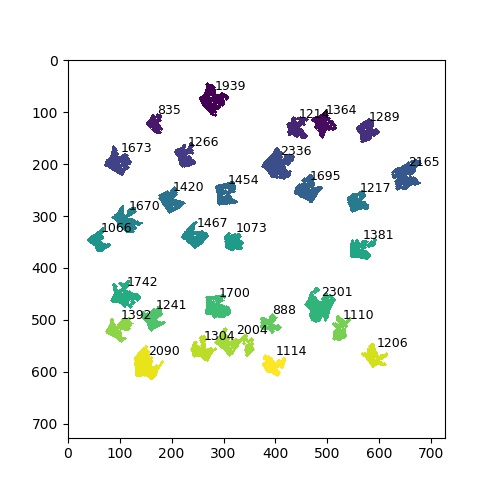
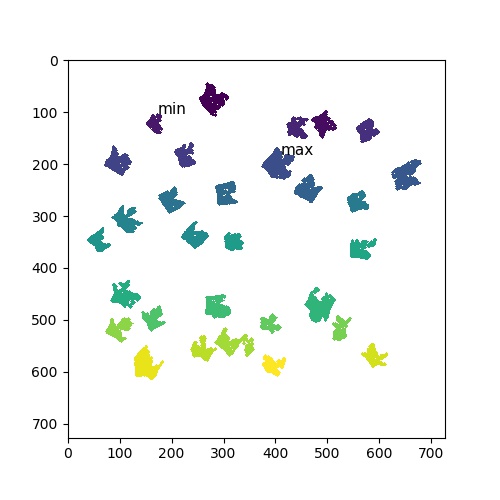
大きさが最大のものと最小のものは以下のコードで求めることができます。
まとめ
SciPyのndimageモジュールのmeasurements関数とmorphology関数を使用することで、サボテンの実生画像から個々の植物を識別し、サイズや形状などの特徴を定量的に計測できることがわかりました。二値化処理とラベリング技術を組み合わせることで、複数のサボテンが写っている画像からでも個体ごとの測定が可能です。この手法は、サボテン栽培における成長記録の自動化や品種分類など、様々な応用が期待できます。
参考

scikit-imageによるラベリング
ndimageより多機能なscikit-imageによるラベリングについて下記で解説した。


コメント
ありがとうございます。
とても勉強になりました。